細胞の直接変換を誘導する転写因子を
高精度で予測するAI手法を開発
― マルチオミクスデータと機械学習を活用した再生医療の開拓 ―
九州工業大学大学院情報工学研究院の山西芳裕教授らの研究グループは、京都大学大学院医学研究科の沖真弥特定准教授との共同研究により、iPS細胞のような未分化細胞を介さずに、すでに分化した細胞を別の種類の細胞へと直接変換するダイレクトリプログラミングを誘導する転写因子を高精度で予測するAI手法を開発しました。
ポイント
- すでに分化した細胞を別の種類の細胞へと直接変換するダイレクトリプログミングを誘導する転写因子を予測するAI手法を開発した。
- 元細胞や標的細胞に関わる生体分子のマルチオミクスデータを融合する機械学習アルゴリズムにより、先行研究よりも高い精度で転写因子を予測することに成功した。
- 今回の提案手法は、今後の再生医療分野における細胞治療のための細胞作製法の開発に繋がることが期待される。
本研究グループは、複数の生体分子情報(マルチオミクスデータ)や細胞間の系統関係情報を統合解析し、すでに分化した細胞を別の種類の細胞へと直接変換するダイレクトリプログラミングを誘導する転写因子を高精度で予測するAI手法を開発しました。ダイレクトリプログラミングを誘導する転写因子を実験的に同定するのは、時間や実験コストの面から非常に困難なため、情報科学的な予測手法が切望されていました。今回の提案手法では、皮膚由来の線維芽細胞を、さまざまな組織(肝臓、骨、脳、心臓、膵臓、腸)由来の細胞へと、それぞれ直接変換させる転写因子を予測しました。さらに、細胞変換メカニズムを考慮し、元細胞や標的細胞に関わる生体分子のマルチオミクスデータや細胞間の系統関係情報を機械学習アルゴリズムで統合することにより、ダイレクトリプログラミングを誘導する転写因子を高精度に予測することにも成功しました。本提案手法により、ダイレクトリプログラミングを誘導する転写因子が未知の細胞変換に対しても、転写因子を新たに予測することができるようになり、今後の再生医療分野における細胞作製法の開発を促進することが期待されます。
本研究成果は、2022年4月12日に英国科学誌「Bioinformatics」のオンライン版で公開されました。研究の詳細は別紙をご参照ください。
<研究の背景と経緯>
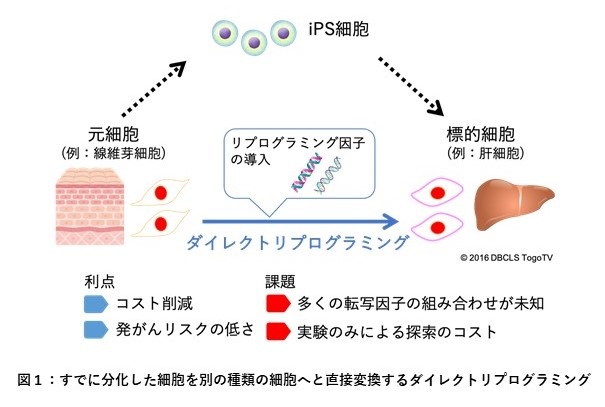
ダイレクトリプログラミングとは、細胞に単独もしくは複数の転写因子※1を導入することで、iPS細胞を経由せずに臓器の機能を持つ細胞に直接変換する過程を指します(図1)。iPS細胞を経由した細胞の作製法より短期間で低コストに細胞を作製することができることから、転写因子の導入によるダイレクトリプログラミング誘導法の研究が注目されており、将来の再生医療を担う革新的な技術として期待されています。
ダイレクトリプログラミングを誘導する転写因子の組み合わせを同定する上で、細胞実験や動物実験のみでは多大なコストと時間を要するため、近年の計測技術向上により蓄積されているさまざまな生体分子情報(マルチオミクスデータ)を活用したデータ駆動型による転写因子の予測手法が切望されています。しかしながら、ダイレクトリプログラミングの分子生物学的機序に基づいた予測手法は未だに確立されておらず、また、予測結果の信頼性や精度は未だに乏しいことが課題となっていました。
<研究の内容>
九州工業大学大学院情報工学研究院大学院生(当時)の江口凌平、濱野桃子助教、岩田通夫博士研究員、大学院生(当時)の中村透、山西芳裕教授らの研究グループは、京都大学大学院医学研究科の沖真弥特定准教授との共同研究により、ダイレクトリプログラミングを誘導する転写因子を高精度に予測するAI手法を開発しました。
本研究では、細胞変換に関与するエピジェネティック※2な修飾の変化を介して転写※3を活性化するパイオニア因子※4に着目し、クロマチン構造※5から標的の細胞種それぞれのパイオニア因子を推定しました。次いで、様々なマルチオミクス情報や細胞間の系統関係情報を統合し、パイオニア因子を考慮した上でダイレクトリプログラミングを誘導する転写因子を予測する世界初のコンセプトを提唱しました (図2)。
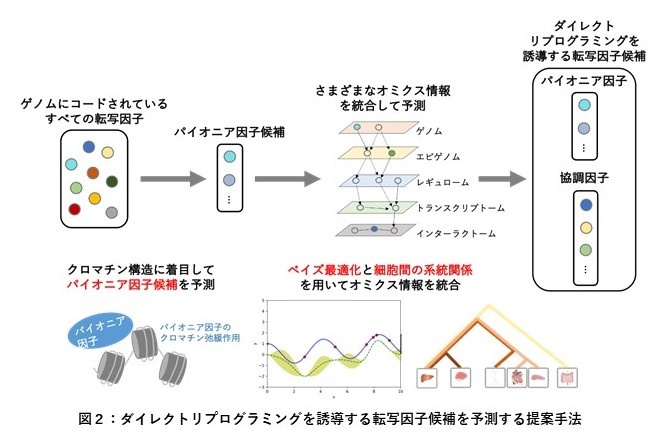
1.ゲノムにコードされているすべての転写因子から、元細胞から標的細胞に直接変換する転写因子候補を予測する。まず、クロマチン構造に着目し、凝集したクロマチン構造を弛緩する機能をもつパイオニア因子を推定する。
2.元細胞や標的細胞に関するマルチオミクス情報や細胞間の系統関係情報を用いて、協調因子(パイオニア因子と協調して働く転写因子)を推定し、最終的に転写因子候補セットを予測する。(マルチオミクス情報と細胞間の系統関係情報を統合するため、ベイズ最適化を用いた機械学習アルゴリズムを開発した。)
本提案手法を用いて、線維芽細胞から肝臓、骨、脳、心臓、膵臓、腸の6種類の細胞への直接変換を誘導する転写因子を予測しました(図3)。その結果、全ての標的細胞に対して転写因子の予測を行うことができ、また、予測された転写因子にはパイオニア因子を含む転写因子の組み合わせが同定されました。予測された転写因子には、先行研究でダイレクトリプログラミングを誘導することが実験的に証明されている既知の転写因子を多く含んでおり、予測の妥当性を確認できました。例えば、線維芽細胞から肝細胞への直接変換では、予測上位10個のうち既知の転写因子6個を正確に再現できました。このことから、本提案手法を適用することで、既知の転写因子を含むダイレクトリプログラミングを誘導する転写因子の予測が可能であることが明らかになりました。
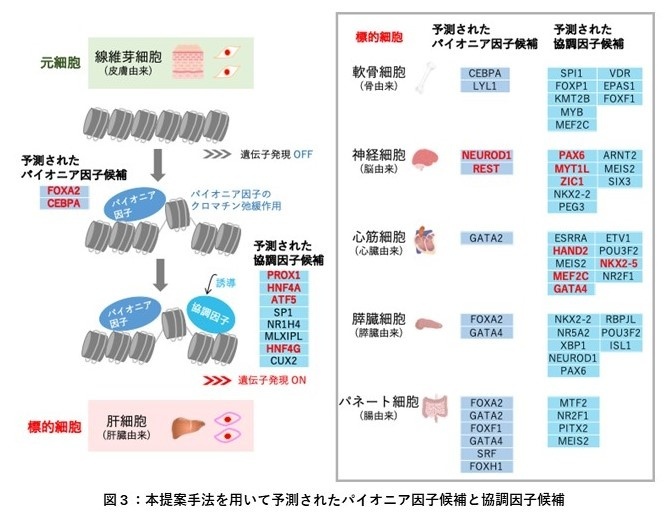
線維芽細胞からさまざまな標的細胞への直接変換を誘導するパイオニア因子と協調因子の候補を示す。パイオニア因子は、凝集したクロマチンを弛緩させ、協調因子の結合を誘導する。赤字で示す転写因子は、先行研究でダイレクトリプログラミングを誘導することが実験的に証明されている転写因子を示す。
次いで、本提案手法と3つの先行研究手法の予測精度の比較を行いました。予測スコアの高い上位10個の転写因子のうち、各標的細胞に対して実験的にダイレクトリプログラミングを誘導することが証明されている転写因子の数を比較しました (図4)。その結果、本提案手法は、3つの先行研究手法より多くの既知の転写因子を再現することができ、最大で67%の精度向上を達成するなど、高い予測精度を確認しました。
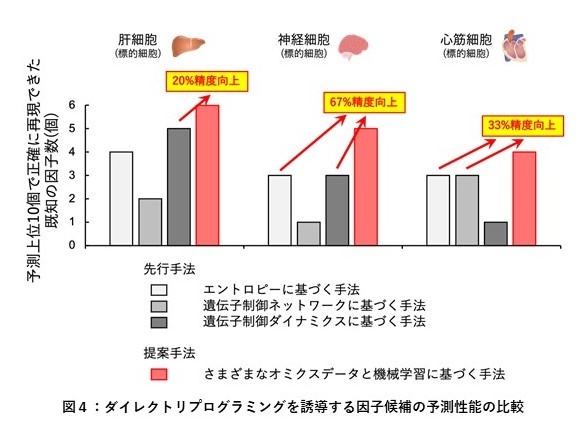
比較のため、エントロピーに基づく手法(Entropy-based method; D’Alessio et al., Stem Cell Reports, 2015)、遺伝子制御ネットワークに基づく手法(Mogrify; Rackham et al., Nat. Genet., 2016)、遺伝子制御ダイナミクスに基づく手法(DGC-based method; Ronquist et al., Proc. Natl. Acad. Sci., 2017)を用いた。本提案手法を用いることにより、線維芽細胞からさまざまな標的細胞への直接変換に必要な転写因子を、より精度良く予測することができる。
<今後の展開>
本研究では、ダイレクトリプログラミングを誘導する転写因子を精度良く予測するAI手法を開発しました。本提案手法は、細胞の変換過程において重要な役割を果たすパイオニア因子を考慮している点が特長であり、マルチオミクスデータ(ヒストン修飾、遺伝子発現、ChIP-seq、エンハンサー領域、分子ネットワークなど)と細胞間の系統関係情報をベイズ最適化などの機械学習アルゴリズムで統合した先駆的な情報技術です。
本提案手法は、元細胞と標的細胞のデータさえ用意できれば任意の細胞変換を扱うことできるため、ダイレクトリプログラミングを誘導する転写因子の組み合わせが同定されていない細胞変換への応用も可能です。このことから、本研究は再生医療における細胞治療法の開発への貢献に繋がることが期待されます。
本研究は、日本学術振興会?科学研究費助成事業?挑戦的萌芽研究における研究課題「低分子化合物による転写因子制御と細胞直接変換のためのインシリコ予測法の開発」(研究代表者:山西芳裕)、挑戦的研究(開拓)「AIによるデータ駆動型ダイレクトリプログラミングの創生と腫瘍化リスクの回避」(研究代表者:山西芳裕)の支援を受け行われました。
<用語解説>
※1 転写因子
DNA に結合して遺伝子の発現を調節するタンパク質であり、調整する標的の遺伝子に応じて多様な種類、組み合わせがある。
※2 エピジェネティクス
DNAやDNAが巻きついているヒストンタンパク質の化学的修飾により、DNAの塩基配列の変化を伴わずに遺伝子発現が制御される仕組みのことを指す。
※3転写
遺伝子の発現を調節するために、DNAからRNAが写し取られる過程のことを指す。
※4 パイオニア因子
高度に密集したクロマチン構造に結合してそれを弛緩させ、抑制されていた遺伝子発現を活性化する状態を作りうる特殊な転写因子のことを指す。
※5クロマチン構造
ヒストンタンパク質8量体にDNAが約150塩基対巻きついたヌクレオソームを基本構造とし、その形状を動的に変えることで遺伝子の発現を制御している。
■ 発表雑誌
| 論文タイトル | “TRANSDIRE : data-driven direct reprogramming by a pioneer factor-guided trans-omics approach” |
| 著者 | Eguchi, R., Hamano, M., Iwata, M., Nakamura, T., Oki, S., and Yamanishi, Y. |
| 雑誌名 | 「Bioinformatics」(オンライン版:04月12日) |
| DOI | https://doi.org/10.1093/bioinformatics/btac209 |
【研究内容に関するお問い合わせ】
九州工業大学大学院情報工学研究院
生命化学情報工学研究系 教授 山西芳裕
TEL:0948-29-7821
E-mail: yamani*bio.kyutech.ac.jp
【取材報道に関するお問い合わせ】
国立大学法人 九州工業大学 広報課広報係
TEL:093-884-3008
E-mail: sou-kouhou*jimu.kyutech.ac.jp
(メールは*を@に変えてお送りください)